在高分辨质谱分析领域,偏生物样本的代谢组学与偏环境样本的非目的分析都是近期比较火的研究方向。这里关注的待测物一般是分子量1500Da或2000Da以下的小分子,相比蛋白质组学而言看起来是简单了但其实分析逻辑复杂度更高。从定性角度来说,基于质谱的定性要依赖精确质量数与其分子碎片的特异性,蛋白质组学的麻烦在于分子量大了以后精确质量数测定受同位素与多电荷影响谱图解析会有困难,实践里都是打碎了测肽段,然后利用算法重新拼出蛋白质序列,当然自上而下的测序现在也有些新思路,更多依赖仪器进步。到了小分子部分,蛋白质组学里存在的同位素与多电荷问题相对少一点,但这里就不存在肽段的概念了,也就是小分子测定里没有类似碱基或氨基酸的结构单元,此时单纯依赖高分辨质谱只能得到分子式,但要想知道结构,必须要有碎片信息。
这里我先明确下为什么我不用所谓“非靶标”来翻译non-target 或untargeted 这个词。首先非靶标跟非目的就不是一回事,目的分析更多针对研究目的,靶标更多侧重已知物,前者是研究概念而后者是分析概念。在具体研究中,应该使用目的而非靶标,例如我想找一种已知物的代谢物,已知物我们有标准品而代谢物没有,那么这算靶标还是非靶标?没有标准算非靶标的话事实上你又知道其与已知物可能就存在一个质量差,也正是利用质量差来找代谢物;算靶标你又没有标准,只是有个筛选概念。但如果直接算目的性分析就没有问题,因为我研究目的就是代谢物,真正的非目的分析应该是完全意外的发现或基于效应引导分析的发现,例如田振宇老师对6PPD-quinone的发现。
现在很多人名义上说是非目的分析,实际做的却是全扫描配合DDA/DIA之后去跟质谱数据库做比对。坦白说这跟你买了标准在自己实验室搞个本地数据库做比对从结果上看区别不大,后者无论如何也不能叫做非目的分析,因为逻辑上你根本就不打算测数据库之外的物质,你的目的实际上是讨论已知数据库里的物质。在我看来,能在研究开始前就得到标准谱图的物质都不能算到非目的分析里,只是高阶版目的分析或筛选,基于已知代谢物或污染物的下游分析例如代谢通路分析事实上都是在对已有的研究进行低质量验证工作,结论都只能基于已知代谢通路来讨论,谈不上有什么新发现。当然,新发现从来都是很困难的,能把已知通路影响做出来也算不错,目前很多人对真正非目的分析的可行性表示了怀疑,更多去关注已知通路的评价,也就是事实上做的都是目的性分析。这样的结论从分析角度更可靠,但似乎除了展示些花里胡哨的系统性影响外也很难有新假说提出,做的结果大都是看上去高端大气但读了也不知道具体在说啥,其他学科的人也始终对此持怀疑态度而应用受限。
我觉得这个问题还是有解的,非目的分析关注的重点应该是信息收集而不是物质鉴定。如果你关注物质鉴定,那么肯定掉到数据库匹配的目的性分析老路上。但如果关注信息收集而把物质鉴定剥离出去,思路就会很明确,那就是最大限度收集样品里的小分子信息。看上去跟物质鉴定一样,但实际操作是不一样的,因为你需要保存的结果不是鉴定出的物质列表而是可能的谱图,前者是一个终点而后者可以反复去跟更新的数据库匹配来进行探索发现工作。举例而言就是你说我测到了一万个峰,鉴定出了一百个物质,但我更关心的是这一万个峰里不论有没有名字究竟有多少物质。很多所谓鉴定物质只有个名字而没有任何文献报道其功能,实质上跟没鉴定出来区别不大,你给他起名字叫物质XYZ都可以,真实研究关注的是其分子功能,但这个很多非目的分析的文章反而不去讨论,只去关注可鉴定物质间的关系,结果大概率还是其他通路研究的一个远程验证。
那么具体在质谱上如何操作呢?第一步毫无疑问是全扫描,这里只说软电离部分,硬电离属于后面分子碎片的讨论。理论上软电离全扫描拿到的应该是物质的分子离子峰,但理论跟现实的距离好比卖家秀跟买家秀,看上去相似但其实根本不是一回事。即便是最常见的ESI源,单个物质软电离下会同时出现同位素峰、加合物峰、中性丢失峰还有一些莫名其妙的寡聚体与各种峰的多电荷峰。我下载了HMDB的LC-MS质谱数据,算了下大概平均每个物质的所谓MS1标准谱图也有26个峰,而GC-MS这种硬电离的平均峰数为90。这就是现实跟理论的差距,LC-MS的全扫描软电离数据里甚至还有碎片离子峰可以用来做定性,相关研究可以查阅广州工业大学薛靖川老师的工作。
但我们的目的是收集物质信息,从信息角度一个物质我只需要一个峰,其余的峰给的信息都是冗余的。只要我找到单一物质的一个峰且最好是分子离子峰,那么我把这个峰送去做二级质谱得到结构信息,那么也就完成了信息收集。也就是说,如果我对每一个潜在物质找到一个峰,那么实际上样品里所有可以测到的物质信息我就都可以收集到二级谱图了,哪怕现在鉴定不了,起码我也可以根据谱图相似性做功能性推测。这也就是标题所说的“竭泽而渔”式非目的分析。
那么怎么去掉冗余峰呢?常见思路就是给分别找出同位素峰与加合物,然后排除掉,这里就需要一个事先定义的质量差列表。不过我前面也说了,这还是目的性分析的思路,利用经验法则来排除。但真实样品里加合物是非常复杂的,套用已知质量差的列表会引入假阳性,例如样品里本没有某种加合物,但你去套这个列表就会误把不同的物质鉴定为加合物关系。我的思路很简单,既然只关注样品里存在的加合物,那么我就去算样品里的单独保留时间区间里的质量差,然后计算下各种质量差的出现频率,频率高的就标注为潜在加合物,这样就降低了假阳性的概率。其实,在真实样品中经常会发现很多不常见的高频率质量差,但也不用过度解释,直接归为未知加合物就可以了。然后,通过峰强度的相关性计算出潜在加合物间的相关性峰网络,这个网络大概率就来自于同一物质。之后,我们就可以根据一些自定义的规则来选择其中的一个峰作为潜在的先导离子,例如选强度最高的。这样我们就把全扫描的数据处理成了独立物质峰数据,一个峰代表一个物质,算是“竭泽而渔”,有些研究人员则称为伪目的分析(pseudo targeted analysis)。从我经验而看,如果20分钟全扫描可以得到一万个峰,那么独立物质峰个数大概只有一千这个量级。
那么是不是这一千个峰就可以用目的性分析的方式作为先导离子来采集二级质谱呢?如果只进样一次是不行的,因为PRM模式下20分钟扫一千个先导离子灵敏度完全不够,二级谱质量非常差。这里需要一个技巧,那就是根据先导离子的保留时间写一个多次进样脚本,让每次进样都测定样品中一部分互相不干扰灵敏度的先导离子。从我经验而言,一千个先导离子大概需要10次重复进样就可以覆盖所有独立物质峰。这样在样品进样阶段,我们就收集了所有可能的物质结构信息(二级谱图),至于啥时候能鉴定出来,这个一来可以等后面出现更全的数据库,二来其实对很多研究而言,给个名字并不重要,例如我们发现很多物质都是甲氧基化的,那么可以直接去验证相关酶的活性而不是去费力研究所有甲氧基化物质具体是什么。生物信息重要的是过程而不是名字。
前面介绍的就是我最近发表在 Journal of Cheminformatics 上的PMDDA工作流,整体流程如下:
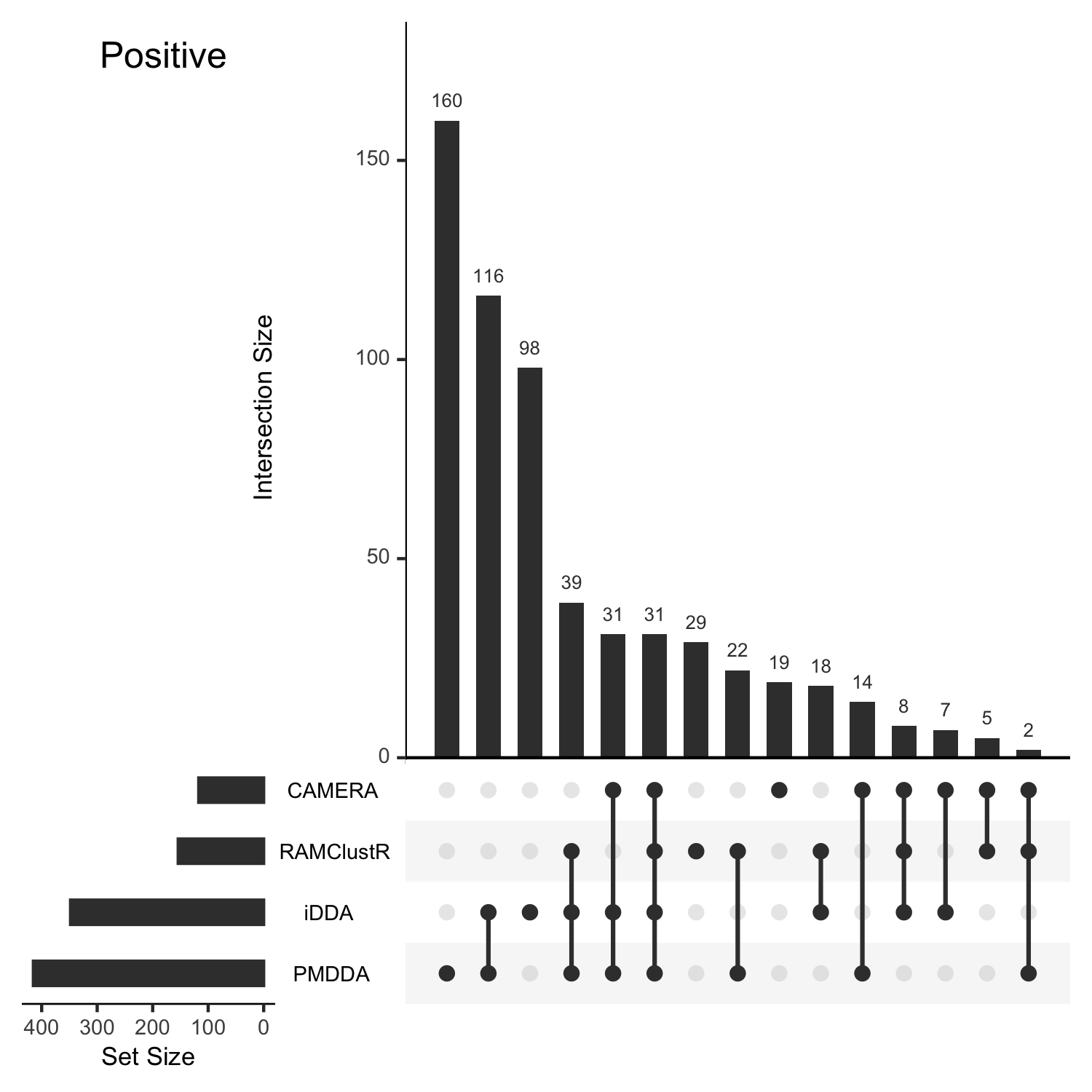
那么效果如何呢?我比对了利用已知加合物质量差列表的CAMERA或RAMClustR,这两个软件都有提供MS1谱的功能,可以直接给出潜在物质的精确质量数,我就用这些数据计算出先导离子荷质比并同样进行重复进样。最后我同时对比了PMDDA/CAMERA/RAMClustR 选择先导离子后采集的二级谱图在GNPS上的分子网络图(可理解为潜在物质)。结果如下图,可以看出这种基于样品自身信息选择先导离子的方法要全面优于基于已知质量差列表的方法。
同时我其实也对比了常见的DDA(一年前的预印本里有,后来因为加入iterative DDA在正式论文里被移除掉了),DDA的问题是牺牲了灵敏度去采集二级谱图而其先导离子虽然依赖样品自己,但选到的峰其实质量非常差,这就出现了DDA测到的很多物质在MS1数据里压根就找不到的现象。这里我要说下很多现在发表的工作经常把MS1的数据与MS2的数据分开来讨论,其背后的小心思就在于MS2里好不容易发现的能解释得通的物质在MS1里完全找不到或数据不支持结论,这就是DDA滥用的后果。其实DDA倒也可以把MS1里选择的先导离子作为优先离子,但问题是如果你关注的就是MS1里能测到的物质,PRM就是能收集所有信息灵敏度最高的方法了,DDA肯定会把扫描时间浪费在MS1里没有的离子上。我还额外测试了iterative DDA,这项技术需要仪器支持,每次扫描只会扫前一次扫描里没有扫到的先导离子,效果跟PMDDA差不多,但让我比较吃惊的是PMDDA跟iterative DDA测到的MS1里的物质很多是不重叠的。也就是说,如果你真想竭泽而渔,那么最好的方法就是在样本量允许情况下同时进行PMDDA与PMDDA辅助的iterative DDA。
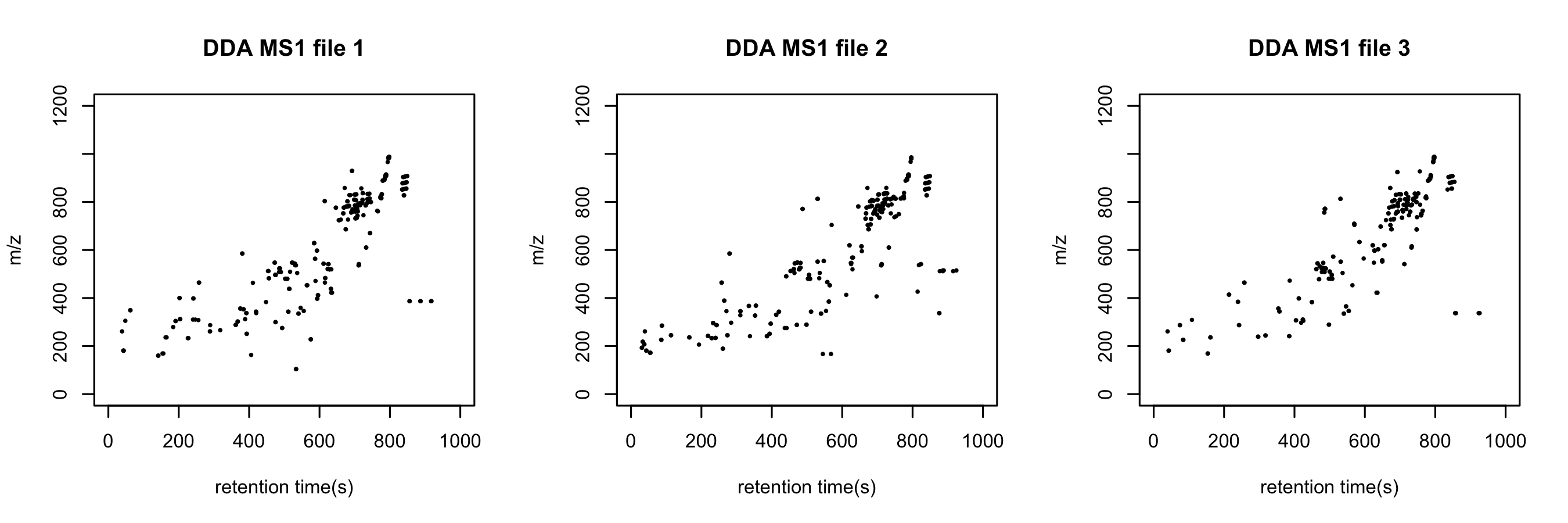
这里我要补充下文章里没详细描述的一部分经验内容,那就是“竭泽而渔”的效果。从独立分子网络或者说潜在化合物的数目上看,即使使用PMDDA,先导离子里有二级谱图生成的也不多,大概只有一半多一些,而DDA收集的二级谱图从数目上看是足够多的,但问题是它选的先导离子并不存在于MS1的峰表里。形成这个现象的原因可能是我的MS1峰列表排除掉了样品中峰响应小于空白中同一峰响应三倍的峰,但进行这个峰过滤的原因在于这样的峰通常峰形很差,不能拿来定量,但DDA却会因为这些峰符合top10的选择原则就给送去做二级了。所以,我很难说“竭泽而渔”的PMDDA一定能拿到比DDA更多的二级谱图,但从其与MS1质量较高的峰表的对应效果而言还是有优势的。而且,我还发现传统DDA的重复进样可重现性非常差,下图展示了三次DDA进样同一样本时其选择的先导离子,基本一针一个样,这使得我更怀疑所有用DDA作出的研究成果了。有意思的是仪器厂家的软件根本就不提供这个功能,所以做科研必须要用开源软件且具备自己验证的能力,否则就是仪器厂家的免费甚至付费复读机。
PMDDA的另一优点在于不需要仪器配合,你完全可以用三重四极杆质谱来采集二级谱信息。这里的关键就是要通过数据分析提取MS1全扫数据通过多次进样来采集高质量的二级谱信息。不过,大连化物所许国旺老师也有类似思路的研究,这里最大区别就在于先导离子的选择策略。此外我对于研究的可重复性要求非常高,研究里用的样品是标准参考物质,数据可以直接从网上下载,所有处理代码是开源的,除了在xcmsrocker镜像里有模版脚本外,我还在GitHub上建了一个仓库保存了本项研究中所有分析细节,包括所有论文里图片的生成过程,而论文在预印本期间就已被多次引用。实际上我现在所在的实验室就用的PMDDA工作流来给其他课题组测样,可靠的工作流要经得起公开透明的各种验证并方便迁移到其他人的研究环境里,但很遗憾很多研究看着很好却因为过渡依赖商用软件很难实现本地化与验证。
如果有问题或对此感兴趣也欢迎随时联系我,我会提供技术支持。