这个问题反复掉坑,在这总结下,省的记吃不记打。
所谓网络可视化,一定是有节点与节点间连线组成,节点一般指代一个样品或特性,连线则代表了样品间或特性间的关系。也就是说,网络的最小单元就是一个两点连线,也就是起点与终点,虽然描述一个网络很直观,但具体到数据结构上就存在一些问题。常规样本数据一般是每一行代表一个样品,每一列代表一个描述样品的维度,样品或维度间的关系并不能展示在原始数据结构里,所以我们需要将样品-维度的数据框转成描述网络的起点-终点数据结构才好可视化。但事实上,你更应该需要一种描述网络的数据类型,然后根据类型定义可视化方法,也就是将原始数据转为网络数据类型,这种类型定义也方便了除可视化外其他针对网络的分析方法开发与使用。
在 R 中,有两个包提供了描述网络的基础类型定义,一个是 network包,另一个是igraph包。这两个包允许用户生成一个专门描述网络的对象,也定义了该对象类型的绘图方法,也就是说,如果你可以直接 plot 一个网络对象,实现快速可视化。很多网络可视化的工具,例如 ggnetwork包或ggraph包或GGally包的ggnet2函数,都支持输入的对象为 network包或igraph包里定义的网络类型。不过,这里面ggraph包还可以基于tidygraph包使用tbl_graph对象来描述网络关系,几乎完全覆盖了igraph包的内容,当然,装这个就得装 tidyverse全家桶。
不论哪一种对象类型,网络对象一定可以抽象出两张表,一张表保存节点的属性,另一张表保存节点间连线的属性。而对于可视化而言,节点属性表其实就是原始数据框,而连线属性表则要保存我们计算出的节点间关系,例如节点间相关性、距离等。然后很自然每一种对象类型都设计了独立的针对节点与边的赋值方法,有了这些方法就可以自定义一些节点或边的属性方便可视化。不过,这里很多人手头只有数据框,也就是只有节点那张表,关系的表还是要自己生成的,在图论里这叫做邻接矩阵(adjacency network)。最简单就是一个相关矩阵或距离矩阵,不过也不一定就是方阵或三角阵,这里面坑多我就不展开说了,多数可视化包都支持你导入一个邻接矩阵或者更原始的起点终点数据框来生成网络对象,然后你可以对节点与边进行属性定义,在可视化时指定需要可视化的属性就可以了。很多人(其实就是我)卡在第一步数据导入就放弃了,但过了第一步能生成网络对象后后面就特别容易进行后续分析。当然,至于说网络可视化的具体布局,其实是存在一些预先设定好的美观布局的,你可以根据需求进行调整,但不要误导读者,做好图例。
这里特别提一下qgraph包,这个包几乎依赖了上面所有提过的包,但这在应用学科中并不少见,例如这个包主要是为心理测量学设计的。但很有意思的是,如果你去搜索 R 语言的网络可视化教程,基本都会找到心理测量学或社会科学背景的人写的东西,而且质量很高,例如Katya Ognyanova的博客,Sacha Epskamp 的博客,Claudia van Borkulo 的博客还有这里,特别最后一个总结并追踪相当多近些年网络科学的主题。打个比方,通常我们说节点间的关系,一般就是想到相关性,但两个节点间也可以用是否独立来构建联系而相关只是独立与否的一种,偏相关行不行?或者如果计算二元而非连续特征值(社会科学里定量研究常用)间的独立性就需要用到 Ising 模型。同时构建出的网络是不是稳定也需要正则化例如 lasso 或重采样来对变量间关系进行调整,去掉不稳定的联系。另外,如何检测一个网络中的社群?有哪些算法?其实背后也是潜在变量分析的影子,这些主题在心理学领域被挖得很深。
如果跳到生物信息学领域,有一个 WGCNA包用的特别多,但据我观察很多写教程的人都没搞清楚原理与模型假设。WGCNA包是基于巴拉巴西的无尺度网络构建的,基本原理就是先对所有基因构建两两相关性矩阵,然后从相关性矩阵中探索出共表达的基因模块,相当于把几千维的基因降维到不到十个且最好能联系上生物学意义例如某个通路啥的,具体计算则是每个模块进行主成分分析(其实是SVD分解),然后用第一个主成分作为这个模块的代表对你的研究分组进行差异分析,找出哪个模块有影响然后解释。这里面核心步骤里有两个坑,第一个在相关性矩阵到基因模块这里,第二个在主成分分析那边。第一个坑是因为其探索模块用了无尺度网络的假设,首先得去选一个幂级数来计算邻接矩阵,这个幂级数是拟合无尺度网络的度分布搞出来的,很多数据本身不符合无尺度网络的度分布,所以硬套这个假设是不合适的。第二个坑跟第一个有关系,只有模块内部第一个主成分可解释方差很高才能这么用,但由于第一个坑很多人用了默认值,第二个坑也就只用了第一个主成分,很多时候方差解释连三分之一都不到,虽然能讲故事,但明显是有偏的。当然这个包里也是涵盖了很多对于用户而言天书级的概念,很多人不求甚解套默认值也把文章给发了,完全就当神奇降维盒子在用了。其实说白了网络分析是另一层意义上的因子分析,起一个降维作用,只是降维方式不是简单的线性组合而是引入了图论的一些统计量罢了,但我看到很多人用起来套代码,解释上完全就是胡说八道,特别是代谢组学里会出现套基因组学的分析方法而不验证假设盲目追求自动化。不过我也看到了很多基于图论的生物信息学文章,很多想法非常超前但引用非常少且真正生产数据的人基本看不懂或不看,这就还不如心理测量学那边研究人员的学科内科普做得好。
说个题外话,其实我能知道很多包的问题不是跟开发者打过交道,而是我查过很多包的源码,非统计与计算机科学背景开发者写的包其实是沉默的大多数,他们一般只会用基础R包函数来实现自己想要的功能,如果没有就会去依赖其他包,很少用 Rcpp,不开并行计算,基本不关心速度,用S3 对象而不是S4,这倒是科研编程的日常状态。有时候读他们的源码有种见字如面的感觉:有的人明显是其他语言转过来的,有次读一个包的代码怎么看怎么别扭,后来发现这个开发者的母语是 java,很多定义方式都是那边传过来的。有的人注释掉的代码其实有另一重意思,源码里保留了很多进化遗迹,类似化石。有的人严谨,每个函数都写测试,文档明显打磨过语言。有的人飘逸,通篇找不到注释,很多编码风格都不一致,感觉是爆栈网复制过来的。有的人很明显是tidyverse 风格出现后才开始学的 R ,对基础函数用法非常不熟。非统计与计算机科学开发者的代码通常存在很多不严谨的地方,没有经过软件工程的训练,更多是为了解决特定目的而快速实现的,不过很多代码展示的想象力非常丰富多彩。
好了,说这么多还是要给点最直观的例子。下面我就手工生成几个网络并做下基础可视化,这里我不会用最常见的那种起点终点数据结构,因为这个东西是需要从原始数据生成的,很少有原始数据本身就是这种关系结构,而且此处我也不涉及 ggplot2 风格的绘图包,用基础绘图系统来做,函数统一为plot,当然不同的对象类型会有不同的绘图参数。
network 版
set.seed(110)
library(network)
# 生成一个3节点网络
net <- network.initialize(3)
# 画出来
plot(net,vertex.cex=10)
# 添加一条边
add.edge(net,2,3)
# 画出来
plot(net,vertex.cex=10, displaylabels=T)
# 添加两个点
add.vertices(net,2)
# 画出来
plot(net,vertex.cex=10, displaylabels=T)
# 模拟一个5*12的数据框
df <- matrix(rnorm(60),5)
# 用邻接矩阵直接生成网络
dfcor <- cor(df)
# 去掉低相关性边
dfcor[dfcor<0.5] <- 0
netcor <- as.network(dfcor,matrix.type = 'adjacency')
plot(netcor)
# 增加节点/边属性
set.vertex.attribute(netcor, "class", length(netcor$val):1)
set.edge.attribute(netcor,"color",length(netcor$mel):1)
# 可视化属性
plot(netcor,vertex.cex=5,vertex.col=get.vertex.attribute(netcor,"class"),edge.col=get.edge.attribute(netcor,'color'))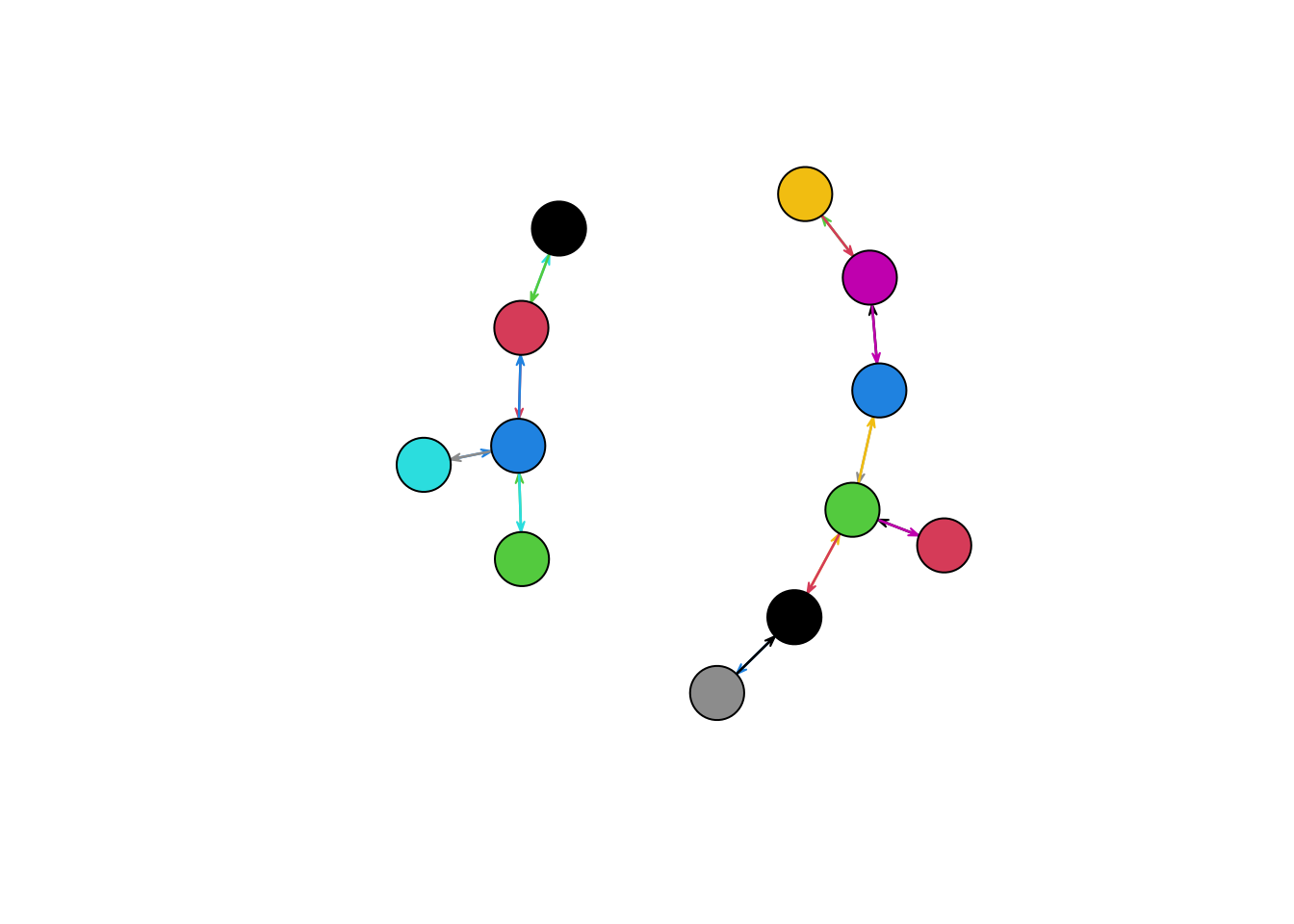
igraph 版
set.seed(110)
library(igraph)
# 生成一个3节点网络
net <- graph.empty(n=3, directed=TRUE)
# 画出来
plot(net)
# 添加两条边
new_edges <- c(1,3, 2,3)
net <- add.edges(net, new_edges)
# 画出来
plot(net)
# 添加两个点
net <- add.vertices(net, 2)
# 画出来
plot(net)
# 模拟一个5*12的数据框
df <- matrix(rnorm(60),5)
# 用邻接矩阵直接生成网络
dfcor <- cor(df)
# 去掉低相关性边
dfcor[dfcor<0.5] <- 0
net <- graph.adjacency(dfcor,weighted=TRUE,diag=FALSE)
plot(net)
# 增加节点/边属性
V(net)$name <- letters[1:vcount(net)]
E(net)$color <- "red"
E(net)[ weight < 0.7 ]$width <- 2
E(net)[ weight < 0.7 ]$color <- "green"
# 可视化属性
plot(net)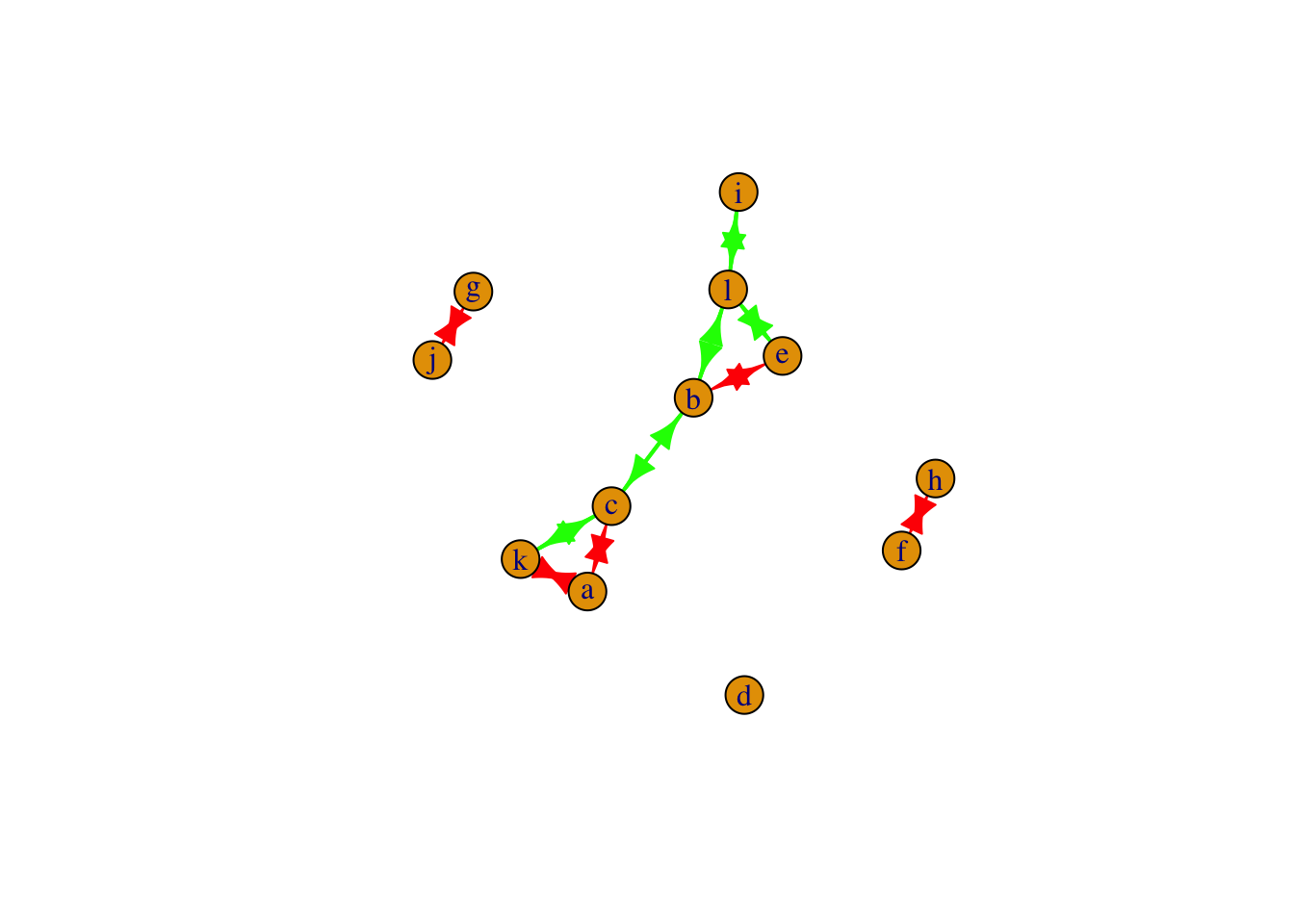
网络可视化只是网络分析的基础,很多基于网络稳定性分析还有网络群组分析都是可以基于更基础的概率图模型来进行,这些分析都有明确的背景问题来源,但涉及的知识点非常多,从统计物理到图论到随机过程,不过如果你带着自己的问题去探索,总会有新的发现。